Journal of the Korean Biblia Society for Library and Information Science 2024 KCI Impact Factor : 1.0
-
pISSN : 1229-2435 / eISSN : 2799-4767
- https://journal.kci.go.kr/kbiblia
pISSN : 1229-2435 / eISSN : 2799-4767
Inferring Undiscovered Public Knowledge by Using Text Mining Analysis and Main Path Analysis: The Case of the Gene-Protein ‘brings_about’ Chains of Pancreatic Cancer
Ahn Hyerim 1, Min Song 2, Heo Go Eun 2
1연세대학교 일반대학원 문헌정보학과
2연세대학교
정보 기술의 발전과 함께 폭발적으로 증가하고 있는 정보 자원을 처리하기 위해 발전한 텍스트마이닝은 방대한 양의 텍스트 데이터에서 자동적으로 유의미한 정보들을 추출할 수 있도록 하는 기술이다. 특히 Swanson의 연구
췌장암은 그 원인이 밝혀지지 않았고, 다른 암에 비해 암 발생의 원인으로 작용하는 암 전단계의 병변 역시 뚜렷하지 않아 조기 진단이 어려우며, 5년 생존율이 5% 이하로 예후가 무척 나쁜 암이다(서울대학교병원 의학정보). 이 때문에 췌장암의 발병 원인 및 그 과정을 규명하기 위한 연구가 다양하게 진행되고 있지만
이에 본 연구에서는 텍스트마이닝과 주경로 분석(main path analysis)을
문헌 기반 발견은 명시적으로 연결되지 않은 문헌에 숨겨져 있었던 새로운 지식을 밝혀내는 과정으로, 미발견 공공 지식 추론이라 칭하기도 한다. 문헌 기반 발견을 처음으로 제안하였던
Swanson이 ABC 모델을 기반으로 수립한 가설이 임상의학자들에 의해 실제 유효한 것으로 검증되면서
이중 그래프를 기반으로 하는 연구에서는 최근 A와 C 사이에 존재하는 중간 개념인 B를한 가지로 제한하는 ABC 모델의 한계를 극복 하려는 시도가 이어지고 있다. Wilkowski 등 (2011)은 처음으로 B개념을 단일 개념이 아닌 여러 종류의 개념 집합으로 확장하여 구체화하였고,
본 연구에서는 B개념의 다수준 모델에 반영 되는 동사의 추출 과정에서 방향성을 반영하여 미발견 공공 지식 추론의 정확성을 높이고, 주경로 분석을 통해 가장 의미 있는 경로를 검출 하려 하였다. 생물학 네트워크(bio network)에서 방향성은 생물학적 프로세스의 역학을 이해 하고 네트워크 분석을 통한 예측을 강화해준다는 측면에서 중요한 의미를 가진다
주경로 분석은 인용 네트워크에서 가장 의미 있는 경로를 추적할 수 있게 하는 네트워크 분석 방법으로, 일반적으로 특정 학문 분야의 발전 궤적을 추적하는 데 활용된다
주경로 분석에서는 내향 연결정도(indegree) 가 0인 모든 노드를 경로의 시작점인 소스(source), 외향 연결정도(outdegree)가 0인 모든 노드를 경로의 끝점인 싱크(sink)라 가정하고 소스와 싱크 사이의 모든 노드와 아크에 관해 횡단 가중치(traversal weight)를 계산한 뒤 횡단 가중치가 가장 높은 경로를 주 경로를 결정한다
본 연구에서는 다음과 같은 두 가지 이유에 따라 유전자-단백질 상호작용 네트워크를 주경로 분석으로 분석하였다. 첫째, 서론에서 언급한 바와 같이 췌장암은 그 원인이나 병변이 뚜렷하지 않으므로 특정 유전자나 단백질을 소스나 싱크로 특정하여 경로를 추론할 수 없다. 따라서 네트워크 내에서 가능한 모든 (즉, 내향 연결정도가 0이거나 외향 연결정도가 0인) 소스나 싱크에 대해 모든 경로를 반영하는 주경로 분석이 대안이 될 수 있다. 둘째, 인용 네트워크에서의 인용의 누적이 해당 학문 영역에서의 영향력을 보여주듯
본 연구에서는 췌장암의 주요 유전자-단백질 상호작용 사슬이라는 미발견 공공 지식을 추론 하기 위하여 (1) 관련 논문을 수집하고, (2) 수집한 논문 전문을 전처리한 후, (3) 개체를 추출 하고, (4) 추출된 개체간의 관계성을 추출한 뒤, (5) 개체와 개체간의 관계성을 종합하여 상호 작용 네트워크를 구축하였다. 이에 대한 상세 설명은 다음에서 순서대로 기술한다.
데이터 수집은 PMC Central에서 이루어졌다. 2014년 10월 기준으로 최근 10년 이내 출판 논문중 제목에 “pancreatic cancer”가 포함되어 있는 논문을 검색하였고, 이 중 전문이 제공되는 1,032건을 다운로드하여 분석에 활용하였다.
수집한 논문 전문의 전처리는 Stanford CoreNLP를 기반으로 일부 코드를 수정하여 진행하였고 UC Denver biolemmatizer, Lucene analyzer 등의 텍스트 처리 관련 자바 오픈 소스도 활용하였다. 각 논문의 전문은 문장 단위로 분할한 뒤불용어(stopword, 523개 적용)를 삭제하였고, 품사 태깅 후 표제어 복원(lemmatization)을 수행 하였다.
개체 추출(named entity recognition)은 사전을 기반으로 하였다. 먼저 유전자와 단백질의 추출을 위해 각각의 동의어 사전(synonym dictionary)을 작성하고 개체 추출에 적용하였다. 두 사전의 개요는
<표 1>. 유전자-단백질 동의어 사전 개요

사전 검색은 최대 8개 단어로 구성된 구 (phrase)에 대해 실시하였는데 해당 구에 품사 태깅이 되어 있으면 품사가 명사일 때만 사전을 검색하였다. 유사도 계산에는 TFIDF(Term Frequency Inverse Document Frequency) 가중치의 일종인 Soft TFIDF 알고리즘을 사용하였다(SecondString Project). 한계치는 0.99로 두고, 검색 결과가 중복 존재할 경우에는 점수가 더 높은 쪽을 선택하였다.
개체 간의 관계 추출은 387개 생물학 동사(bio verb)를 기반으로 진행하였으며, 다음과 같은 단순 규칙을 적용하였다.
•동사는 개체 사이에 위치
•부정문이 아닐 때에만 관계 저장
•품사 태그를 통해 수동태 확인
또한 하나의 동사를 중심으로 좌우에 출현하는 모든 주체(subject) 개체와 객체(object) 개체에 대해 관계를 저장하였다.
본 연구에서는 네트워크의 분석에 네트워크 분석 도구인 Pajek을 활용하였다. Pajek에서는 네트워크를 확장자 ‘net’ 형식의 파일로 저장하는데, 먼저 노드를 나열하고 이후 노드 사이에 존재하는 아크 또는 엣지만을 나열하여 밀도가 낮은 대용량 네트워크의 분석에 적합하다. 본 연구에서는 앞선 단계에서 추출된 개체를 노드로, 개체간의 관계를 아크로 나열하여 ‘net’ 형식의 파일로 저장하고 Pajek에서 네트워크 분석을 실시하였다.
유전자/단백질 개체 및 관계 추출 결과 파싱 중 에러가 발생한 43건을 제외하고 총 989건의 논문에 대해 중복을 제외한 10,068개의 관계가 추출되었다. 관계에 포함된 개체의 수는 1,769 개였다.
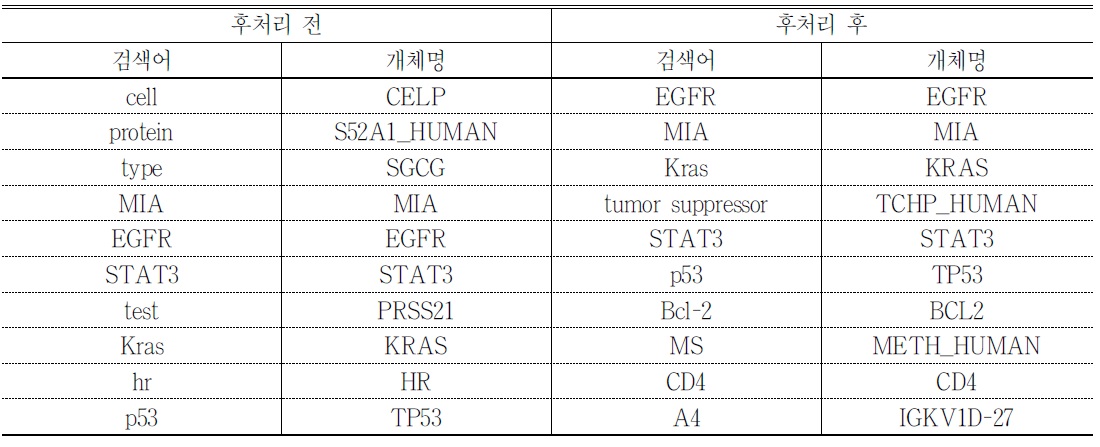
개체 및 관계 추출 결과 검출빈도 상위에 오른 개체들은
<표 2>. 후처리 전후 검출 빈도 상위 10개 개체
•소문자만으로 표기된 한 단어짜리 일반명사는 개체가 아닌 일반명사로 간주한다.
•대문자 알파벳 약어보다 숫자가 먼저 출현하는 구는 개체가 아니라고 간주한다.
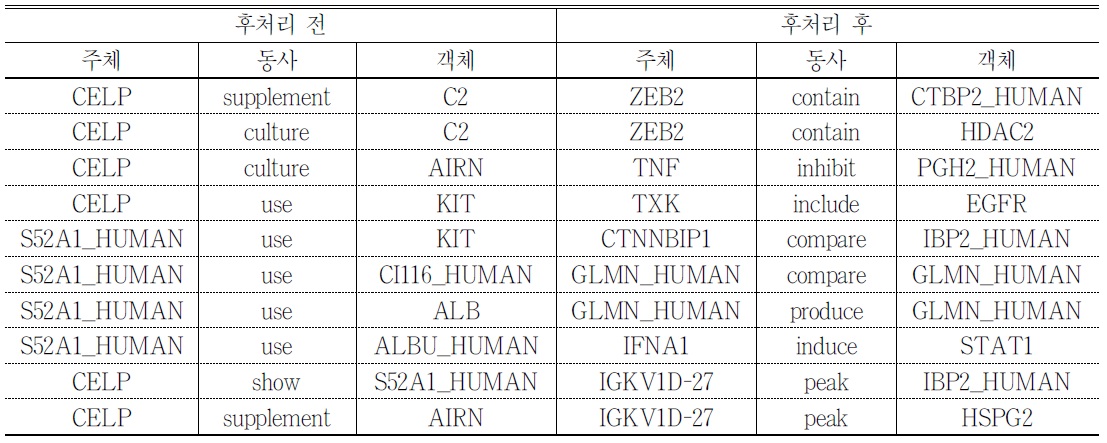
후처리 결과 개체의 수는 1,341개, 이 개체들이 포함된 관계의 수는 중복을 제외하고 5,256개로 감소하였다. 검출 빈도에 따른 상위 10개의 개체와 관계를 후처리 전후로 비교하면 각각
<표 3>. 후처리 전후 검출 빈도 상위 10개 relation
일반적인 네트워크 분석에서는 한 종류의 아크 혹은 엣지를 가정한다. 네트워크에서 아크 혹은 엣지는 노드 간의 관계를 나타내는데, 가중치 혹은 화살표의 방향으로 그 차이를 반영하는 경우도 있지만 일반적으로는 한 네트워크에서 유사한 관계만을 나타낸다. 그러나 본 프로 젝트에서 관계 추출을 위해 사용한 생물학 동 사들은 그 의미가 다양하므로, 관계 추출에서 검출된 모든 관계를 하나의 네트워크로 구성해 서는 분석이 쉽지 않다.
따라서 본 연구에서는 분석을 위해 의미가 유사한 동사를 그룹으로 묶고 이런 동사들이 포함되어 있는 관계를 한 종류의 관계로 가정하여 네트워크를 구성하기로 하였다. 생물학 관계 (bio relation)의 분류에는 UMLS의 관계 구분을 적용하였다(NIH). 단, UMLS의 관계 구분 중 ‘affects’는 긍정적인 영향을 미치는 관계인 ‘positive’와 부정적인 영향을 미치는 관계인 ‘negative’로 추가 분류하였다. 관계 추출에 적용한 387개의 생물학 동사를 분류한 결과는 다음과 같았다.
•functionally_related_to - affects: 65개 (positive: 32개, negative: 33개)
•functionally_related_to - brings_about: 17개
•functionally_related_to - indicates: 11개
•functionally_related_to - performs: 294개
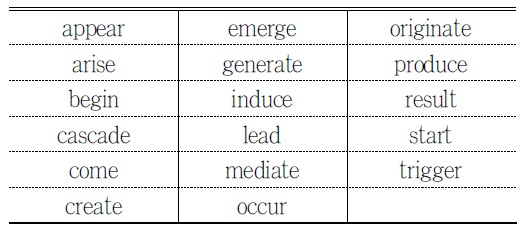
본 연구의 분석 대상이 되는 유발 관계에 포함되는 생물학 동사 17개는
<표 4>. bring_about으로 분류된 17개 생물학 동사
후처리를 마친 5,256개의 관계 중
<표 5>. 췌장암의 gene-protein 유발관계 네트워크 개요

<그림 1>. 췌장암의 gene-protein 유발관계 네트워크
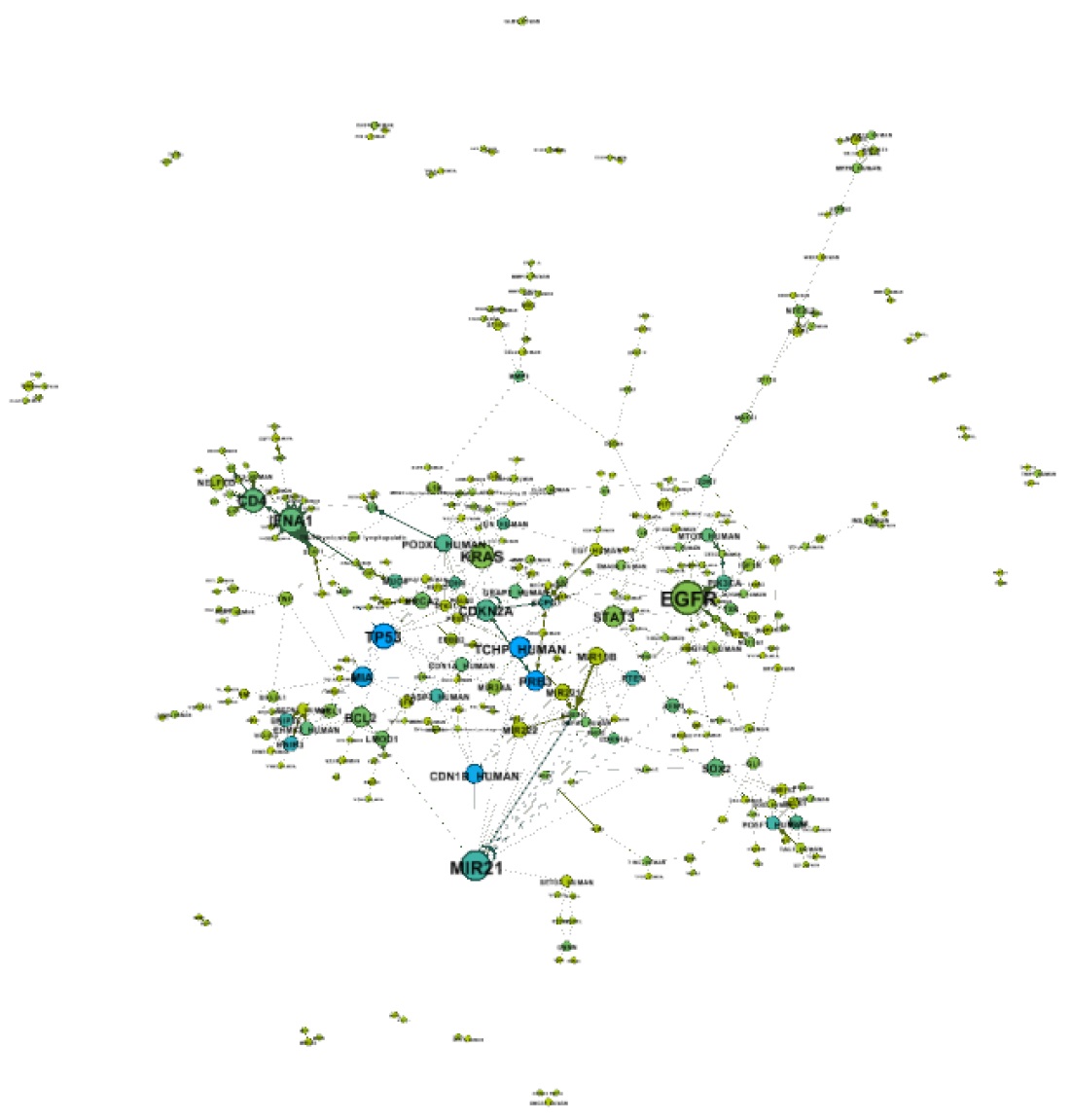
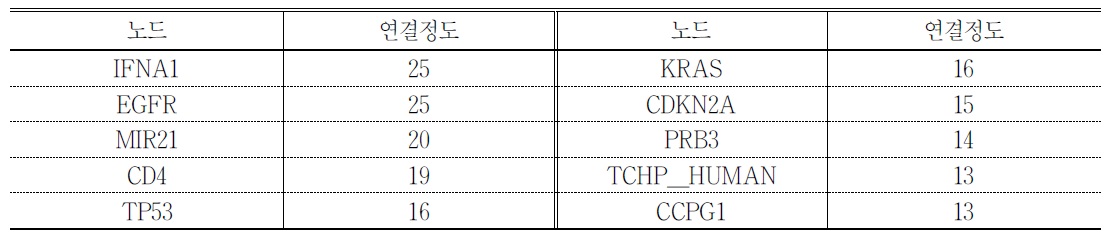
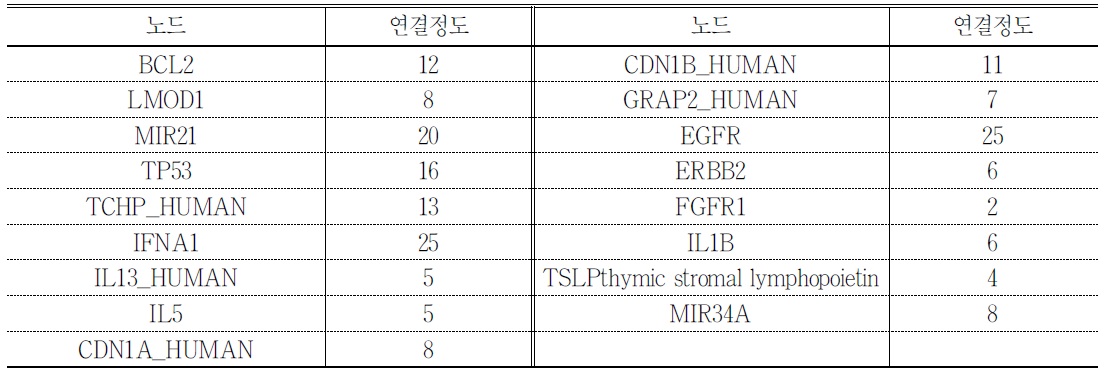
췌장암의 유전자-단백질 유발관계 네트워크는 총 20개의 약한 컴포넌트(weak component)로 구성되어 있으며 이 중 가장 큰 약한 컴포넌트에 288개의 노드가 속해 있다. 연결정도(degree) 기준 상위 10개 노드는
<표 6>. 유전자-단백질 유발관계 네트워크의 연결정도 기준 상위 10개 노드
4.3절에 구성한 췌장암의 유전자-단백질 유발관계 네트워크는 사이클(cycle)이 존재하는 순환 네트워크(cyclic network)이므로 네트워크 내의 사이클을 제거하여 비순환 네트워크 (acyclic network)로 변환한 뒤 주경로 분석을 실시하였다. 순환 네트워크를 비순환 네트워크로 변환하는 과정에서 네트워크의 노드 및 아크의 변화를
<표 7>. 네트워크 변환 과정에서 네트워크의 변화

<표 8>. 네트워크 변환 과정에서 삭제된 node 목록
이렇게 얻은 비순환 유전자-단백질 유발관계 네트워크에서 주경로 분석을 실시한 결과
<그림 2>. gene-protein 유발관계 네트워크의 주경로
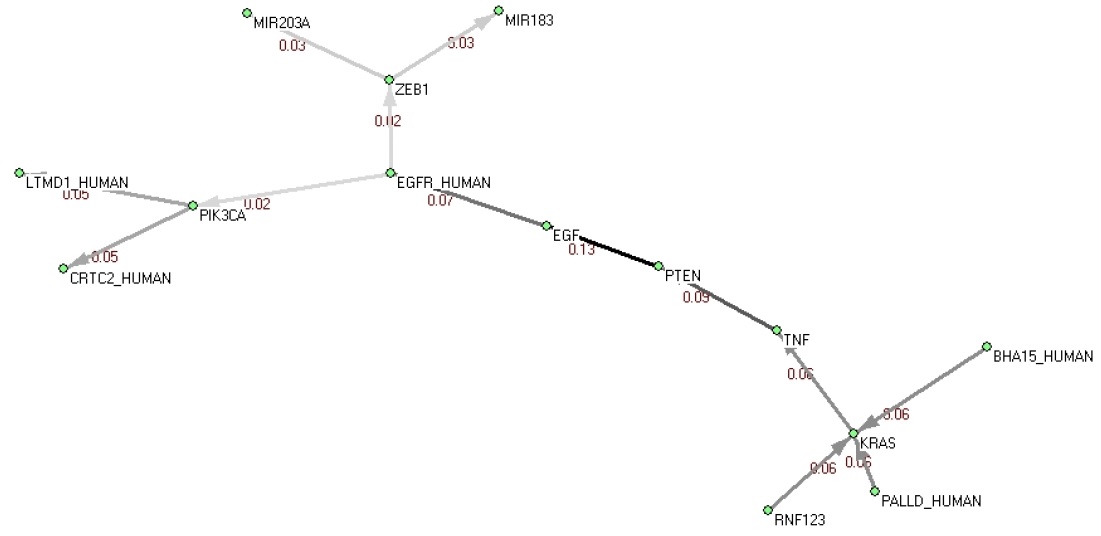
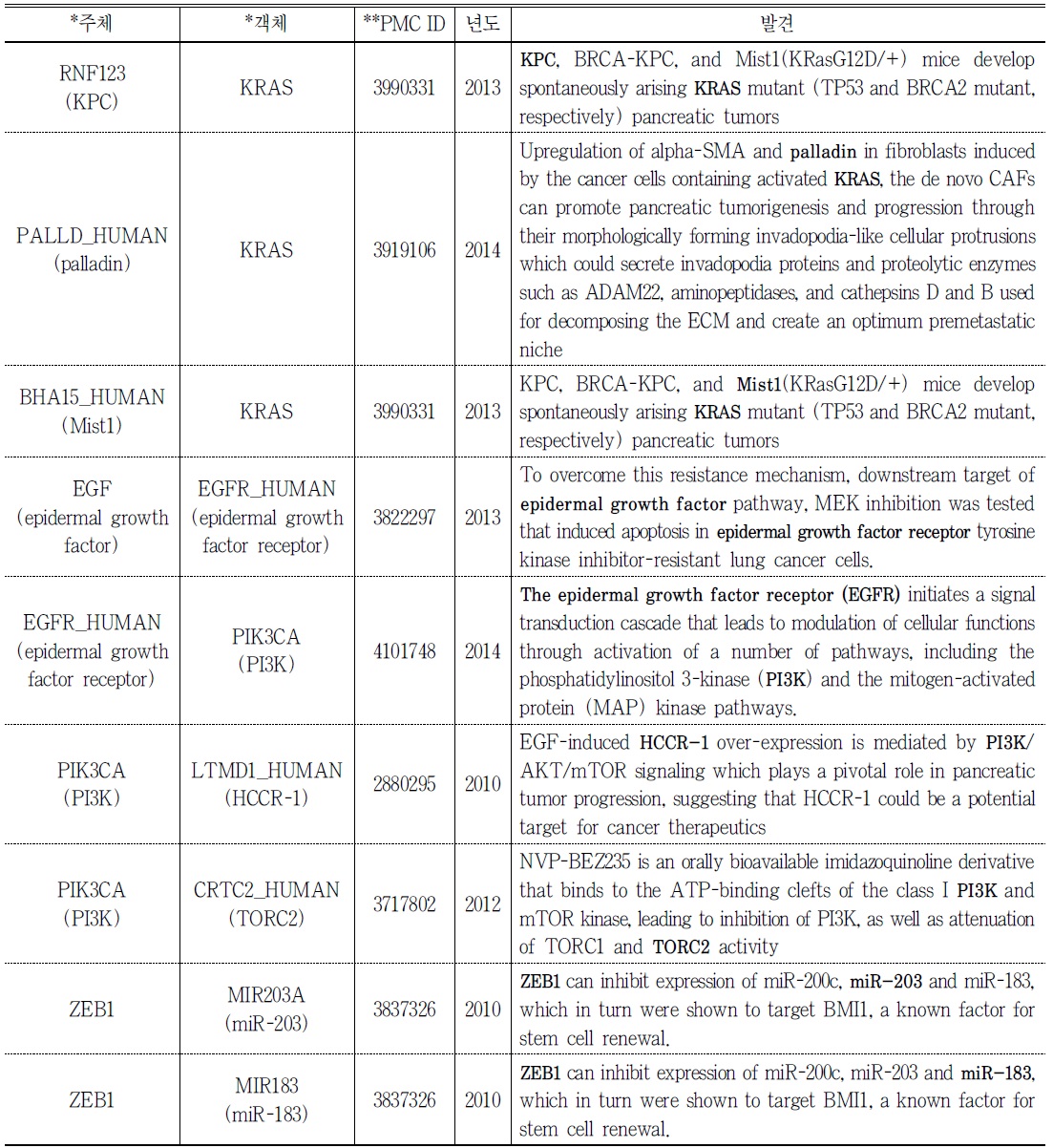
유전자-단백질 유발관계 네트워크의 주경로 에는 14개의 노드와 13개의 아크가 포함되어 있다. 주경로의 아크에 해당되는 13개의 유발 관계 중 개체/관계 추출에서 검출된 관계 9개를
<표 9>. 개체/관계 추출에서 검출된 관계
 * 괄호 안은 검색시 매칭되었던 유의어
** 부록에 PMC ID 순으로 저자 및 제목, 출처를 밝힘
* 괄호 안은 검색시 매칭되었던 유의어
** 부록에 PMC ID 순으로 저자 및 제목, 출처를 밝힘
주경로에 포함되어 있는 14개 노드 사이의 직접 외향 연결 관계를 사이클을 제거하기 전의 유전자-단백질 유발관계 네트워크에서 확인 한 결과는
<표 10>. 주경로에 포함된 노드 사이의 직접 외향 연결 관계
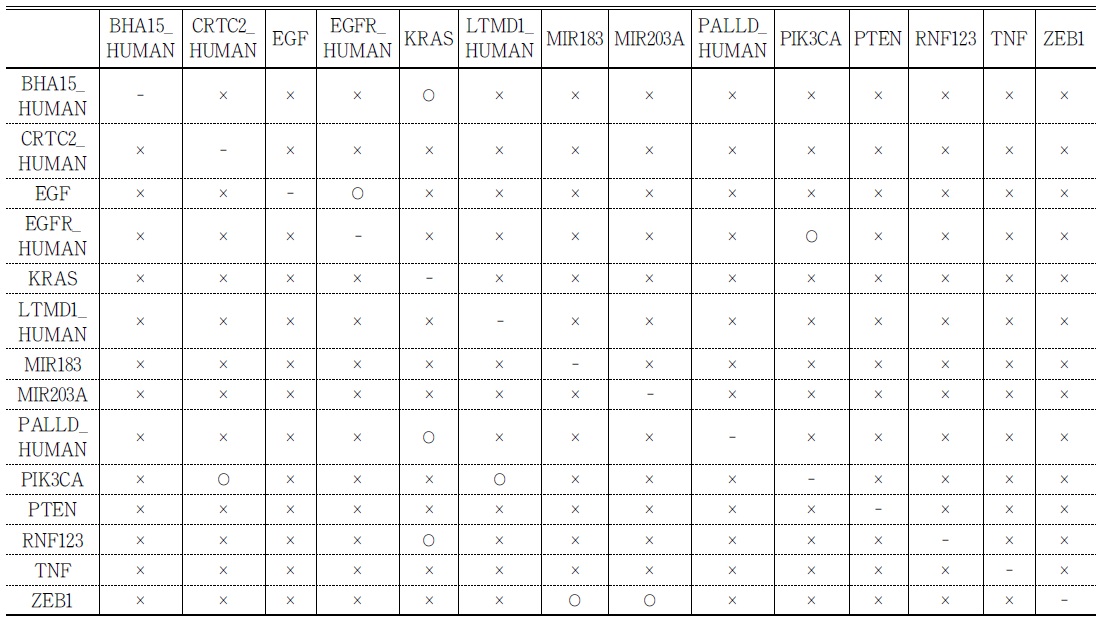 * 두 노드 사이에 직접 외향 연결이 존재하면 ○, 존재하지 않으면 ×로 표기
* 두 노드 사이에 직접 외향 연결이 존재하면 ○, 존재하지 않으면 ×로 표기
생의학 영역에 적용되는 바이오 텍스트마이닝은 생의학적 개념들간의 유의미한 연관성을 자동적으로 발견하고 그 문맥과 상호작용 관계를 이해하여 새로운 지식을 발견해 내는 것을 목적으로 한다. 본 연구에서는 췌장암의 발병 원인과 관련된 문헌 기반 미발견 공공 지식을 추론하기 위하여 텍스트마이닝과 주경로 분석을 Swanson의 ABC 모델에 접목하였다.
텍스트마이닝과 주경로 분석 모두 ABC 모델의 중간 개념인 B를 다단계 모델로 확장하고 가장 의미 있는 개념을 도출하는 데 적용되었다. 최근 10년간 출판된 췌장암 관련 연구 논문을 대상으로 텍스트마이닝을 실시하여 방향성을 반영한 유전자-단백질의 상호작용 네트워크를 구성하였으며, 생물학 동사를 기준으로 생물학 관계를 분류하여 유발관계 네트워크를 분리하였고, 주경로 분석을 통해 네트워크 내에서 가능한 모든 경로 중 주요 유발관계 사슬을 파악하였다.
그 결과 14개의 노드와 13개의 아크가 포함된 췌장암의 유전자-단백질 주요 유발관계 사슬이 검출되었다. 이 중 9개 아크는 실제 관련 연구에 출현한 관계를 반영하고 있었고, 나머지 4개 아크는 유발관계 네트워크를 순환 네트워크에서 비순환 네트워크로 변환하면서 생성된 것으로 확인되었다. 주경로에 포함된 14개 노드 사이에는 주경로에 포함된 연결 관계 외에 다른 연결 관계가 존재하지 않아 검출된 주경로가 우회경로가 아닌 것도 알 수 있었다.
본 연구에서 검출된 주경로는 췌장암 관련 연구에서 주요하게 언급되어 온 유전자-단백질 유전사슬로, 새로운 방법론을 통해 이전까지 발견되지 않았던 연결성을 확인할 수 있었다. 문헌에서의 언급 정도가 실제 상호작용의 중요 도와 비례하는지는 실증적인 연구를 통해 검증 되어야 한다. 그러나 본 연구의 주제가 된 췌장 암의 사례처럼 시작점(소스)와 끝점(싱크)조차 한정할 수 없는 미발견 공공 지식의 추론에서 주경로 분석은 유용한 도구가 될 수 있을 것이다.
1.
[web]
2.
[journal]
허, 고은, 송, 민.
2014
![]()
3.
[journal]
Blagosklonny, M. V., Pardee, A. B..
2002
“Unearthing the gems.”
![]()
4.
[journal]
Cameron, D., Bodenreider, O., Yalamanchili, H., Danh, T., Vallabhaneni, S., Thirunarayan, K., Sheth, A. P., Rindflesch, T. C..
2013
“A graph-based recovery and decomposition of swanson’s hypothesis using semantic predications.”
![]()
5.
[book]
De Nooy, W., Mrvar, A., Batagelj, V..
2005
6.
[journal]
DiGiacomo, R. A., Kremer, J. M., Shah, D. M..
1989
“Fish oil dietary supplementation in patients with Raynaud’s phenomenon: A doubleblind, controlled, prospective study.”
![]()
7.
[journal]
Gustafsson, M., Hornquist, M., Lombardi, A..
2005
“Constructing and analyzing a large-scale gene-to-gene regulatory network Lasso-constrained inference and biological validation.”
![]()
8.
[journal]
Liu, J. S., Lu, L. Y. Y..
2011
“An Integrated Approach for Main Path Analysis: Development of the Hirsch Index as an Example.”
![]()
9.
[journal]
Mattiazzi, M., Curk, T., Krizaj, I., Zupan, B., Petrovic, U..
2010
“Inference of the Molecular Mechanism of Action from Genetic Interaction and Gene Expression Data.”
![]()
10.
[journal]
Natarajan, J., Berrar, D., Dubitzky, W., Hack, C., Zhang, Y., Desesa, C., Van Brocklyn, J. R., Bremer, E. G..
2006
“Text mining of full-text journal articles combined with gene expression analysis reveals a relationship between sphingosine-1-phosphate and invasiveness of a glioblastoma cell line.”
![]()
11. [web] Current Relations in the Semantic Network [online]. http://www.nlm.nih.gov/research/umls/META3_current_relations.html
12.
[journal]
Popa, O., Hazkani-Covo, E., Landan, G., Martin, W., Dagan, T..
2011
“Directed networks reveal genomic barriers and DNA repair bypasses to lateral gene transfer among prokaryotes.”
13. [web] http://secondstring.sourceforge.net/javadoc/com/wcohen/ss/SoftTFIDF.html
14.
[journal]
Selga, E., Oleaga, C., Ramirez, S., de Almagro, M. C., Noe, V., Ciudad, C. J..
2009
“Networking of differentially expressed genes in human cancer cells resistant to methotrexate.”
![]()
15.
[journal]
Swanson, D. R..
1986a
“Undiscovered public knowledge.”
![]()
16.
[journal]
Swanson, D. R..
1986b
“Fish oil, Raynaud's syndrome, and undiscovered public knowledge.”
![]()
17.
[journal]
Swanson, D. R..
1988
“Migraine and magnesium: eleven neglected connections.”
![]()
18.
[journal]
Vinayagam, A., Stelzl, U., Foulle, R., Plassmann, S., Zenkner, M., Timm, J., Assmus, H. E., A. ndrade-navarro, AM., Wanker, E. E..
2011
“A directed protein interaction network for investigating intracellular signal transduction.”
![]()
19.
[journal]
Xiang-Yi, He, Yuan, Yao-Zong.
2014
“Advances in pancreatic cancer research: Moving towards early detection.”
![]()
20.
[journal]
Yan, E., Ding, Y., Sugimoto, C. R..
2011
“P-Rank: An Indicator Measuring Prestige in Heterogeneous Scholarly Networks.”
21.
[journal]
Heo, Go Eu, Song, Min.
2014
“Inferring Undiscovered Public Knowledge by Using Text Mining-driven Graph Model.”
![]()
![]()
22. [web] Pancreatic Cancer [online]. http://terms.naver.com/entry.nhn?docId=926898&mobile&cid=51007&categoryId=51007#TABLE_OF_CONTENT1